
|
電子情報通信学会 - IEICE会誌 試し読みサイト
© Copyright IEICE. All rights reserved.
|

|
電子情報通信学会 - IEICE会誌 試し読みサイト
© Copyright IEICE. All rights reserved.
|
ネットワークの数理モデル
小特集 6.
遺伝子ネットワークの離散数理モデル
Discrete Mathematical Models of Genetic Networks
Abstract
細胞内では様々な遺伝子が相互作用しており,その相互作用はネットワークとして表現される.ブーリアンネットワークは遺伝子ネットワークの代表的な離散数理モデルであり,遺伝子の状態はオンかオフの2値で表され,遺伝子間の制御関係はブール関数で表され,全ての遺伝子の状態が同期して更新される.50年以上前に提案された古典的なモデルであるが現在でも活発に研究されている.本稿では,ブーリアンネットワークと主要研究課題の概要,拡張モデル,生命システムのモデル化事例について説明する.
キーワード:ブーリアンネットワーク,アトラクタ,可制御性,システム生物学,バイオインフォマティクス
細胞内ではDNA,RNA,タンパク質,低分子化合物など様々な物質が相互作用することにより生命を維持している.DNAのうち一部分はタンパク質の設計図となっており,その部分は遺伝子と呼ばれる(注1).タンパク質は遺伝子からメッセンジャーRNAなどを介して生成されるが,遺伝子から対応するタンパク質が生成されている場合,その遺伝子は発現していると言われる.また,発現の程度も一定とは限らず,対応するタンパク質の生成量は遺伝子の発現量と呼ばれる(注2).遺伝子はタンパク質などを介して互いに制御し合って発現量を変化させ,それにより細胞を維持するとともに,外部刺激に対する応答や細胞分裂などの変化をもたらしている.遺伝子間の制御関係を理解することは,細胞や生命をシステムとして理解する上で重要であり,更には,創薬の対象となる遺伝子の発見や病気の治療法の開発にも役立つことが期待される.
遺伝子の制御関係は,遺伝子を頂点に対応付け,という遺伝子が
という遺伝子を制御している場合に
から
への有向辺を引くことによりネットワークとして表現でき,遺伝子ネットワークと呼ばれる.遺伝子の発現量は時間とともに変化するので,遺伝子ネットワークは遺伝子の発現量の動的変化を記述できる必要がある.そのために,様々な微分方程式モデル,差分方程式モデル,ベイジアンモデルなどが用いられてきた.一方,それらのモデルは多数のパラメータが必要であり,全てを正確に記載するのは不可能に近い.そこでネットワークの挙動を定性的に記述できるモデルが必要となる.ブーリアンネットワーク(Boolean Network, BN)はそのために広く研究されている離散数理モデルである(1),(2)(図1).
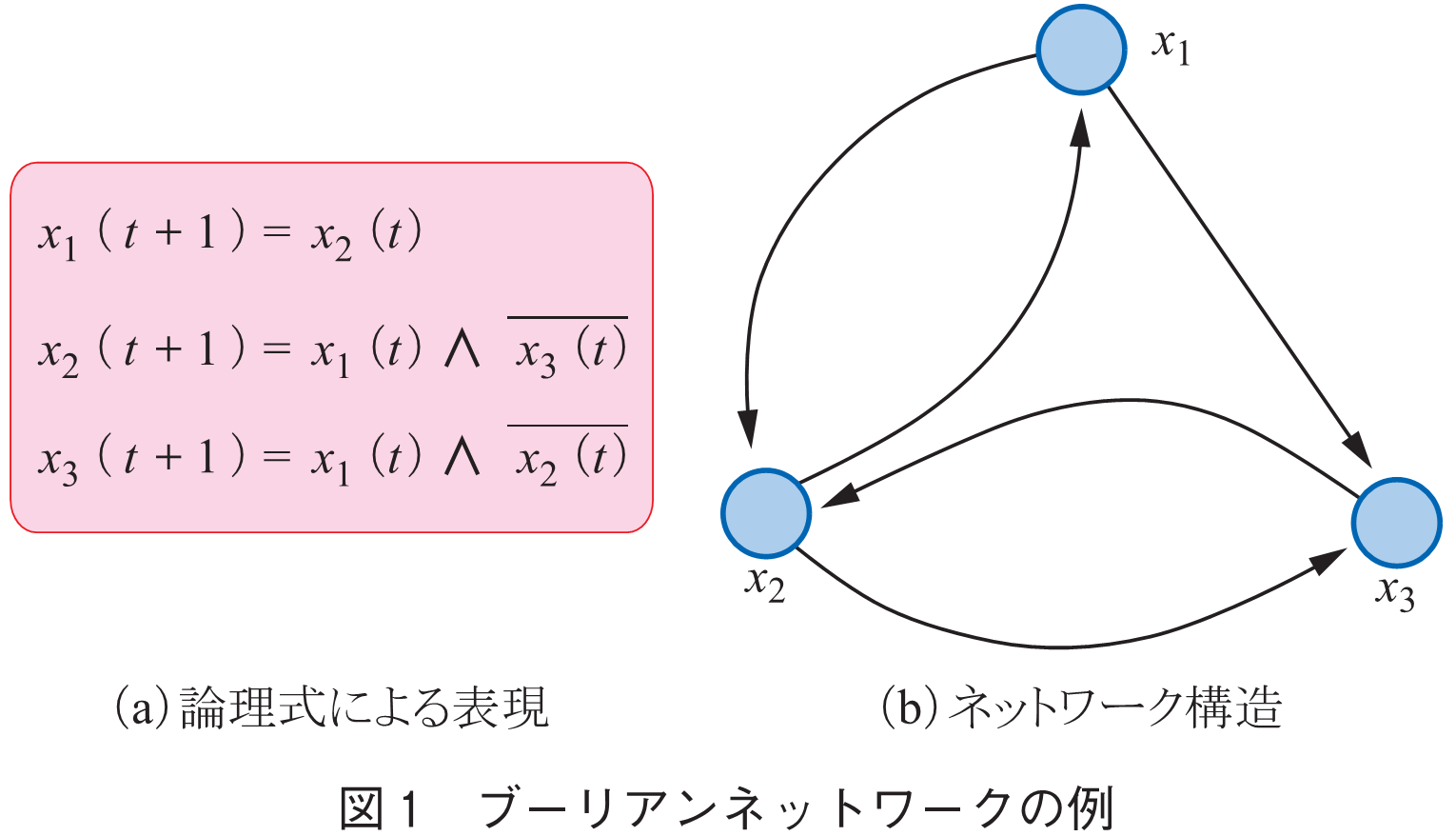
BNでは,頂点が遺伝子に対応し,各頂点は0か1のいずれかの値,すなわち,ブール値をとる.ここで,1は対応する遺伝子が発現していることを意味し,0は発現していないことを意味する.なお,後述するように,モデル化によっては頂点が遺伝子ではなく,タンパク質や化合物,更には,生物種に対応することもある.頂点の状態は単位時間ごとに同期して変化し,その制御規則はブール関数により記述される.0,1の値をとる素子がクロックに同期して変化するのはディジタル回路と同じであり,まさに,遺伝子間の制御関係をディジタル回路として捉えようというものである.BNは1969年にKauffmanにより導入された古典的なモデル(1)であるが,いまだに活発な研究が続いている.更に,数理生物学,統計物理学,バイオインフォマティクス,システム生物学,制御理論など様々な分野の研究者が関わり学際的な様相を呈している.
続きを読みたい方は、以下のリンクより電子情報通信学会の学会誌の購読もしくは学会に入会登録することで読めるようになります。 また、会員になると豊富な豪華特典が付いてきます。
電子情報通信学会 - IEICE会誌はモバイルでお読みいただけます。
電子情報通信学会 - IEICE会誌アプリをダウンロード